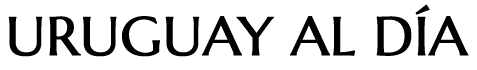MADRID, 13 (EUROPA PRESS)
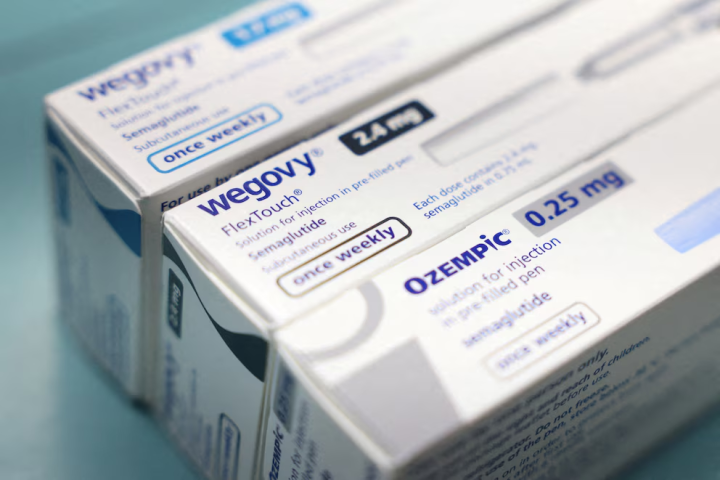
La resistenza antimicrobica (AMR) è una minaccia globale, soprattutto tra i patogeni fungini. Per ottimizzare l'uso degli antimicotici disponibili, i ricercatori del Consiglio Nazionale delle Ricerche spagnolo (CSIC) hanno creato un ampio database e un software per studiare le mutazioni fungine associate alla resistenza antimicrobica.
Per affrontare questa sfida, un team internazionale guidato dall'Istituto di biologia funzionale e genomica (IBFG, CSIC-USAL), dall'Università di Salamanca e dall'Université Laval (Canada), insieme al supporto di varie istituzioni in Canada e nei Paesi Bassi, ha creato FungAMR, un database che raccoglie più di 50.000 voci e 35.000 mutazioni genetiche identificate in 95 specie di funghi di interesse clinico, agricolo e ambientale, associate a 246 proteine e che conferiscono resistenza a un totale di 208 antimicotici.
Queste informazioni sono state estratte manualmente da oltre 500 studi scientifici e rigorosamente classificate in base al livello di evidenza sperimentale, consentendo agli utenti di valutare l'affidabilità e il supporto scientifico di ciascuna mutazione. Il lavoro è stato recentemente pubblicato sulla rivista Nature Microbiology.
"La motivazione alla base dello sviluppo di FungAMR è stata l'urgente necessità di un database completo, dettagliato e affidabile sui meccanismi di resistenza antimicotica. La disponibilità di FungAMR in un'interfaccia web intuitiva e di facile utilizzo ne faciliterà notevolmente l'utilizzo", sottolinea Christian R. Landry, ricercatore presso l'Université Laval.
Inoltre, il team ha sviluppato il software ChroQueTas (Chromosome Query Targets), uno strumento bioinformatico pionieristico che consente l'analisi dei genomi e/o proteomi fungini e il rilevamento automatico delle mutazioni che conferiscono resistenza agli antimicotici. Disponibile come software gratuito, ChroQueTas facilita lo screening genetico rapido, accurato e scalabile di ceppi clinici e ambientali.
"Nei test che abbiamo eseguito sui genomi di studi pubblicati, ChroQueTas ha segnalato in modo affidabile mutazioni precedentemente descritte, oltre a identificare mutazioni non segnalate in precedenza in tali genomi, dimostrando il suo potenziale come strumento chiave per la sorveglianza genomica", afferma Narciso M. Quijada, ricercatore presso l'Institute of Functional Biology and Genomics (IBFG).
Il lavoro rivela che molte mutazioni conferiscono resistenza a più antimicotici contemporaneamente (resistenza crociata), a causa delle loro simili modalità d'azione, e che questa resistenza può derivare dallo stesso meccanismo in specie diverse. L'uso diffuso di antimicotici in agricoltura, in particolare degli azoli, è identificato come una delle principali pressioni evolutive alla base di questo problema. Questa situazione rende difficile il trattamento delle infezioni fungine e sottolinea l'urgente necessità di sviluppare nuove terapie con meccanismi d'azione alternativi.
FungAMR è ora disponibile come interfaccia web all'interno del portale Comprehensive Antibiotic Resistance Database (CARD, https://card.mcmaster.ca/fungamrhome) e verrà aggiornato periodicamente con nuovi dati. Gli utenti possono contattare il database via email all'indirizzo fungamr.db@gmail.com.